Új
lehetőségek a korszerű reumatológiai kutatásban:
genomika és immuninformatika/immunomika
Falus András
az MTA levelező tagja, egyetemi tanár
Semmelweis Egyetem Genetikai, Sejt és Immunbiológiai Intézet, Budapest
MTA–SE Immungenomikai Kutatócsoport – faland @ dgci.sote.hu
Buzás Edit
egyetemi docens, az orvostudományok kandidátusa
Semmelweis Egyetem Genetikai Sejt és Immunbiológiai Intézet, Budapest
buzedi @ dgci.sote.hu
Az orvostudomány ma mindinkább az ún. komplex betegségek felé fordul. Ilyen például az asztma, a kardiovaszkuláris kórképek, a daganatos betegségek, az obesitás kóros formái, és nyilvánvalóan ide tartozik a reumatoid artritisz is. Az autoimmun megbetegedések fejlett diagnosztikája alapján ezen kórképek előfordulási gyakorisága a nyugati társadalmakban 5 %-ra becsülhető, tehát igen jelentős betegségcsoportról van szó. Ugyanakkor a modern orvostudománnyal szembeni egyik legsürgetőbb kihívást éppen az autoimmun megbetegedések mindmáig megoldatlan oki terápiája jelenti.
A genomika eszköztárának felhasználásával igen intenzív kutatómunka folyik világszerte abban a reményben, hogy a fenti, igen komplex megbetegedések esetében a hajlam korai felismerése révén egykor lehetőség nyílhat az autoimmun megbetegedések megelőzésére, illetőleg egyénre szabott terápia kifejlesztésére.
Ennek a hosszú útnak a legelején járva is rendelkezünk számos olyan eredménnyel, melyek az autoimmun megbetegedések közös genomikai sajátosságára hívják fel a figyelmet (Chandonia – Brenner, 2006).
E betegségek lényegében mindegyike poligénes, multifaktoriális, hatalmas populációkat érint, és legtöbbjük az egyes etnikumokban más fenotípust (illetve endofenotípus-kombinációkat) és más genetikai asszociációkat mutat. Az óriási kihívást a tudomány is csak multifunkcionálisan képes kezelni, egyes klinikai, laboratóriumi vagy akár genetikai paraméterek külön csak a legritkább esetekben jelentenek elegendő támaszt.
Ezt a nagyon nagy kihívást látszik kielégíteni a rendszerszemléletű biológia és benne a genomika; a genom alapú, genom léptékű biológia, az emberiség intellektuális teljesítményeinek egyik legkiemelkedőbb sikere a 3. évezred kezdetén, a globalizált tudás előremutató példája. A hatalmas, naponta bővülő, interneten elérhető adatbázisok, a high throughput technológiák (microarray, chip), az elképesztő ütemben fejlődő bioinformatikai eszközrendszer, a multicentrikus tevékenység láncolatába kapcsolt intézmények („gridek”) sokasága forradalmian új minőséget produkál a biológia minden területén.
Az egészséges és beteg szervezet élettani és patológiás folyamatai (hasonlóan szinte minden életjelenséghez) annyira összetettek és annyira szerteágazóan és több szinten (sejten belül, sejtkapcsolatokban, szervek között, a szervezet és a populáció szintjén) reguláltak, hogy a kérdések valódi megközelítésére ma már kizárólag a systems biology (rendszerszemléletű biológia) kísérelhet meg választ adni. Ez a biológiát alapvetően átható rendszerszemlélet egyedi komponensek (molekula, sejt, szerv, szervezet) helyett azok több szinten is regulált hálózataiban gondolkodik, legyenek azok a génkifejeződést szabályozó molekulahálózatok (lásd proteomika, metabolomika) vagy sejtek kapcsolatai. Minthogy ez a terjedő „omics” megközelítés a hálózatok elemeinek aktivitását szimultán vizsgálja, ezeket a jelenségeket csak a nagy teljesítőképességű kvantitatív genomikai technológiákkal lehet követni. Ennek megfelelően könnyen hozzáférhető hatalmas adatmennyiség kiértékelése számítógépekkel történik, mert a jelenségek komplexitását csak informatikai algoritmusok képesek kezelni.
Jó példa a rendszerszemléletű biológia és a bioinformatika előtérbe kerülésére az immunológia mai története, ahol az immunomika kialakulása az immungenomika/proteomika és informatika különlegesen gyümölcsöző egymásra találását láthatjuk. Erre példa az „intelligens” vakcinák szemünk láttára kibontakozó sikertörténete is.
Az eredményalapú virtuális immunrendszereket is igénybe vevő kutatás potenciális gyógyszertargetek sokaságát fogja felszínre hozni az autoimmun, fertőző, gyulladásos, allergiás és egyéb klinikai kihívásokkal kapcsolatban is.
Röviden a genomikáról
A genetika, azaz öröklésbiológia, a vertikális biológiai információátadás tudománya, napjainkban átlépett egy tudománytörténeti küszöböt. A közvetett, majd megfigyeléseken és kísérleteken alapuló „klasszikus” genetika után, a molekuláris biológiai technológiai forradalom nyomán bekövetkezett sok organizmus, többek között az emberi genom nukleotidsorrendjének gyakorlatilag teljes feltárása. Ez a fejlemény azzal vált mára, a harmadik évezred fordulójára korszakalkotóvá, hogy létrehozta az informatika és a biológia valóban tudományos mérföldkőszerű egymásra találását – a bioinformatikát. A genomika nem más, mint genom alapú biológia és annak résztudományai (például onkogenomika, farmakogenomika, immungenomika). Ez azt jelenti, hogy a genomika biológiai összhangzattant jelent, a DNS-szintű variációk és a génexpressziós mintázatok összességének bioinformatikailag értékelt összességét.
A genomikai nagyteljesítményű technológiák által produkált „adatóceán” az emberi gondolkodás történetében eddig nem tapasztalt új helyzetet teremtett. Úgy gondoljuk, nem véletlen, hogy a biológia (orvostudomány, mezőgazdaság) kérdésköre, a megközelítési stratégiák elérték a komplex informatikai megközelítés igényét, hiszen a biológiai rendszerek funkciói és működészavarai csak hálózatokban és azok modelljeivel érthetőek meg. A sejten belüli intracelluláris, a sejtek közötti intercelluláris és a szervrendszerek közti fiziológiás (neuronális, endokrin, immunológiai) hálózatok egyébként rendkívül emlékeztetnek szociológiai hálózati rendszerekhez.
Ez a paradigmaváltás három, tudománytörténeti értelemben egyenként is jelentős előrelépés időbeli és motivációs egybeesése következtében vált reális lehetőséggé.
Az első alap a genomprogramok első, „lexikális” szakaszának befejezése. Mint tudott, 2001 első hónapjai óta már baktériumok, az élesztő, az ecetmuslica, a fonálféreg és egyes növények mellett az emberi genom, legújabban pedig az egér, a kutya lényegében teljes genomiális géntérképe rendelkezésre áll, a világháló adatbázisaiból lehívható és elemezhető. Bár a gének annotációja (azonosítása) még sok időt vesz igénybe, ez a lexikális tudásanyag új genetikai információs minőséget jelent. Ma már sok tucat faj teljes genomját ismerjük, és internetes adatbázisokon el is olvashatjuk.
Emellett óriási és naponta bővülő adatbázist jelentenek a pontmutációk (single nucleotide polymorphism – SNP) tömegének feltárása, hiszen a minden genomban megtalálható milliós nagyságrendű SNP-k egyenként külön és független allélként, genetikai markerekként kerülhetnek felhasználásra. Az emberi genomban mintegy 15 millió SNP található. A mérés technikai kivitelezése ma már nagy kapacitású (high-throughput) rendszerekben is megoldott. A 2002 óta létező hatalmas Hap-Map projekt több millió SNP-t tartalmaz, és szabadon elérhető. A legtöbb haszonnal valószínűleg az SNP sorozatok, mintázatok együttes mérése és detektálása jár majd (de máris vannak biztató eredmények például a Crohn-betegség esetén). Ma már olyan teljes genomot is vizsgálni tudó módszerek is léteznek (például komparatív genom hibridizáció – CGH), mellyel a az egyes szakaszok kópiaszáma is megállapítható.
A második, a nagyteljesítményű microarray (chip) eljárás, melynek speciális jellegzetességei nagyságrendekkel emelik az egyidejűleg vizsgálható gének számát, szerkezeti (nukleotidsorrend) és funkcionális (génkifejeződés-mRNS) információk tömegét képes nyújtani. A génchipek (génlapok) rendezetten (microarray), sorokban és oszlopokban kis felületen is több tízezer ismert nukleotidszálat tartalmaznak, ezekhez kapcsolódik a jelzett minta nukleinsav. A leolvasás letapogató optikai eljárással történik.
Végül a harmadik, a legalapvetőbb, az egész biológiát lényegileg átalakító eszközrendszer a bioinformatika. Ez az új biostatisztikai/biomatematikai megközelítés korrelációs, clusterképző eljárásokkal és egyéb, naponta bővülő szoftvermegoldásokkal elemzi a genomiális/expressziós adatbankok és a génchiptechnika által szolgáltatott adathalmazt, majd biológiai következtetések levonására alkalmas elemzést nyújt. Az egyik legsikeresebb szoftvereljárás az ún. disease pathway elemzés, ahol egyes ismert biológiai aktivációs utakban (például jelátviteli utak, sejtciklus vagy apoptózis utak, vagy akár limfocita-aktiváció) szereplő komponensek génjeinek összefüggő láncolatát, funkcionális klasztereit keresi a program.
A bioinformatika különleges értéke az in silico megközelítés, tehát a nemzetközi adatbázisokhoz kapcsolódó számítógépes munka (dry lab), ahol esetleg laboratóriumi munkától (wet lab) elkülönülten is végezhető korszerű, kreatív, de egyben „génhalászaton” alapuló genomikai kutatás. Ez lehetőség térben (távolról) és időben (régebben) végzett adatgyűjtés értékelésére, és ez teszi a genomikát (együtt a proteomikával, metabolomikával) igazán integratív genomikává. Nagyon izgalmas a szakirodalom elemzésére fejlesztett szoftverek használata, amikor több százezer közlemény szövegelemzése nyomán kialakított „consensus hálózatokra” vetítik egy kísérlet eredményeit, ezzel behelyezve azt a „bibliomikai” hálózatba. Ezen a szinten találkozik az SNP (tehát DNS) mintázat a génexpressziós mintázattal, és képes kiemelni az érintett (esetleg későbbiekben gyógyszertarget) géneket a „betegségútvonalban”. Így tudtak például legújabban a skizofrénia és a bipoláris betegség, illetve az autizmus genetikai mechanizmusaira nézve alapvetően újat mondani.
Az immunológia,
mint kombinatorikus tudomány
A genomika/posztgenomika korszakában, élve az adódó technológiai, informatikai fejlődéssel nem egyes géneket, hanem gén- és géntermékhálózatokat, mintázatokat, komplex útvonalakat, érdemes vizsgálni. Nem véletlen, hogy az elterjedt modellként vizsgált, autoimmunitásra fokozottan hajlamos egerekben vagy transzgenikus állatokban az ún. trait analízis, a funkció (vagy a kóros funkció) kapcsolt gének hálózatára utal, mint például az inzulinhiányos cukorbetegségben (IDDM), reumatoid artritiszben (RA), szisztémás lupusz eritematózuszban (SLE) és pajzsmirigybetegségekben, ami a kórképek humán megfelelőjének poligénes jellege miatt nem is meglepő.
A már ismert gének összefüggéseinek feltárása mellett igazi „génhalászat” is végezhető. Különböző stimulációkra (anti-CD3, anti-CD28, mitogének, ionomicin és kombinációik) például a T-sejten belül eltérő expressziós mintázatok alakulnak ki, ezek analízise révén eddig nem ismert vagy csak más funkciójában ismert gének tucatjait ismerjük fel. Mindez (a klinikai gyakorlatban például) predikcióra ad lehetőséget.
Az immunológus, ideértve az immunbetegségekkel foglalkozó klinikust is, célja a szervezet védekező reakcióinak valamilyen előrebecslése. Itt említhető például a patogénekre, a tumorra, az allergénekre, illetve az autoantigénekre való immunválasz, de természetesen az immunhiányok predikciója is. Korábbi ismereteinket kiegészítve ma már úgy gondoljuk, hogy például a CD4+ helper T-sejtek széles kategóriáján belül az immunválasz kimenetele szempontjából nagyon fontosak a nagyrészt fékező hatású CD4+ CD25+ regulátoros sejtek (Treg). Csak a genomika, az expressziós mintázat globális analízise lesz képes jelentős támpontot adni a CD4+ effektor és a CD4+ regulatorikus sejtek sejtbiológiai különbségére, de feltehetően itt találjuk meg a funkcionális különbség ma még molekulárisan alig feltárt lényegét is. Ilyen eljárással derült ki például, hogy a Foxp3 gén szelektíven fejeződik ki a Treg sejtekben. Ez a predikciós lehetőség ad új, individuálisabb megoldásokat az immunfarmakológia, illetve a vakcináció stratégiája és technológiája számára.
Az immungenomika ma már elterjedten használja az egyes sejtek expressziós mintázatát, a sejtspecifikus immuntranszkriptomot.
Néhány sajátosság, ami alapján az immunológiát kombinatorikus tudománynak tekinthetjük, és ami azt kiemelten alkalmassá teszi informatikai elemzésre:
1. az antigénreceptorok repertoárjait kombinatorikus eseménysor (génátrendeződés és szomatikus hipermutáció) hozza létre
2. a molekuláris és celluláris kölcsönhatások jól vizsgálhatóak
3. komplex genetikai és jelátviteli utak tanulmányozhatóak
4. in vitro (például sejtkultúrákban) és in vivo (például vakcináció követése) kísérletek egyaránt viszonylag könnyen megvalósíthatóak
5. nagyszámú adat keletkezik mérésenként
6. jó modellek (például immunológiai betegségek, transzgenikus állatok) állnak rendelkezésre
7. már ma is nagy betegség-adatbázisok (például MHC, vírusok, autoimmune immunom) léteznek.
Autoimmun betegségek és genomika
Az autoimmun megbetegedések hátterében a saját antigénekkel szembeni kóros immunreaktivitás figyelhető meg. Egy-egy autoimmun kórkép incidenciája önmagában nem magas, de mint betegségcsoport, előfordulási gyakoriságuk jelentős.
Az autoimmun megbetegedések multifaktoriális eredetűek, ez számos gén és környezeti tényező együtthatását feltételezi. Mindössze néhány, kivételt képező autoimmun kórkép esetében áll egyetlen gén mutációja a háttérben, mint például az autoimmun limfoproliferatív szindróma (ALS syndroma) és az autoimmun poliglanduláris endokrinopátia kandidiászisszal és ektodermálisz diszpláziával járó formája (APECED szindróma) esetében. Az autoimmun betegségekre való öröklött hajlam vizsgálata egyike napjaink legkomplexebb genetikai vizsgálati rendszereinek. Az immunrendszeri betegségek háttere eredendően igen összetett (elsősorban a lókuszok nagyfokú alléldiverzitása miatt), melyhez még hozzájárul a szuszceptibilitás poligén jellege.
A ma már komplex módon, szimultán vizsgálható, legfontosabbnak ítélt hajlamosító allélek felelőssé tehetők korábban nem vizsgált allélek együttállásának tanulmányozására a reumatoid artritiszes betegekben és kontrollszemélyekben. Ezáltal lehetőség nyílik egyszersmind az episztázisos génkölcsönhatások (a különböző lókuszok által kódolt molekulák egymással kölcsönhatásban állnak, és egy adott lókusznak megfelelő genotípus befolyásolja a másik lókusznak megfelelő fenotípust) vizsgálatára is, ami a reumatoid artritisz betegséghajlamának kutatásában nem rendelkezik hagyományokkal.
Az autoimmun megbetegedések komplexitása rendszerszemléletű megközelítést igényel. A nagy áteresztőképességű technológiák és számítástechnikai háttér segítségével vizsgálhatók a szabályozó hálózatok dinamikus kölcsönhatásai oly módon, hogy egyidejűleg számos kölcsönható komponens elemzése történik.
Reumatoid artritisz
A reumatoid artritisz multifaktoriális, komplex genetikai hátterű megbetegedés, ez számos gén és környezeti tényező együtthatását feltételezi.
A több évtizedes kutatómunka eredményeképpen mára a kóros autoimmunitásra való genetikai hajlam néhány általános sajátossága ismertté vált.
1. A genetikai hajlam kialakításában a legjelentősebb szerep a fő hisztokompatibilitási rendszer (MHC) génjeinek jut.
2. A szuszceptibilitási lókuszok jelentős mértékben átfednek a különböző autoimmun megbetegedések között (így például a reumatoid artritisz esetében négy, eredetileg idiopátiás diabetes mellitusban (IDDM) azonosított szuszceptibilitási lókusz, az IDDM6, IDDM9, IDDM13 és a DXS998 szerepét igazolták). Ez alapján nem zárható ki annak a lehetősége sem, hogy számos kórkép esetében léteznek közös patogenetikai útvonalak, melyek akár közös terápiás stratégiák célpontjai is lehetnek.
A reumatoid artritiszre való hajlam kialakításában a genetikai tényezők szerepét igazolja, hogy egypetéjű ikrekben a RA konkordanciája 12-15 %, míg kétpetéjű ikrekben 5 % körüli érték. A RA-es populációs variancia kb. 60 %-a genetikai eredetű (Wordsworth et al., 2001; MacGregor et al., 2000).
A genetikai hajlam kialakításában az elsődleges szerep a HLA (humán leukocita antigén) géneké: a teljes genetikai hajlam mintegy 30 %-áért a HLA régió a felelős. A RA-es betegek 65-80 %-ban fordul elő a HLA DRB1* 04 allélhordozás (szemben a kontrollokban megfigyelhető 25-34 %-os gyakorisággal).
Egy közelmúltban végzett egész genomszűrés (whole genome screening) a HLA* DRB1-et a RA fő szuszceptibilitási lókuszaként azonosította (p=0,00004). Az RA jellegzetes összefüggést mutat azon HLA DRB1 allélekkel, melyek a DR1 lánc konzervált, a 70-74. aminosav szekvenciáját, az úgynevezett „shared epitope”-ot kódolják (Gregersen et al., 1987). Ez a szekvenciarészlet a molekula peptidkötő gödrében található. A fenti szekvenciát hordozó allélek a következők: a DRB1*0401, DRB1*0404, DRB1*0405, DRB1*0408, DRB1*0101, DRB1*0102 és a DRB1*1001. A DRB1*0401/*0404 genotípus a korai kezdetre és különösen súlyos lefolyásra hajlamosít. A DRB1*0401/DRX hordozók esetében (ahol az X egy, a „shared epitope”-ot nem kódoló allél), 4,7-szeres a relatív kockázat RA-ra nézve, míg a DRB1*0404/DRX genotípusúak ötszörös, a DRB1*0401/DRB1*0404 heterozigóták esetében pedig 31,3-szoros relatív kockázattal kell számolnunk (Hall et al., 1996).
A HLA DRB1 allélek öröklődése erős kapcsoltságot (linkage disequilibrium) mutat egyes DQB1 allélekkel, mint például a DQB1*0301-gyel (HLA-DQ7) és a DQB1*0302-vel (HLA-DQ8) (Ilonen et al., 1990).
A nem-HLA gének közül igazoltan meghatározó szerepű a RA-re való genetikai hajlam kialakításában az NFKBIL1 lókusz. Ezenkívül meghatározó még az arginin citrullin átalakítást végző peptidyl arginin deimináz enzim PADI4 lókusza (Suzuki et al., 2003), a protein tirozin foszfatáz PTPN8 gén missense SNP-je (Begovich et al., 2004), valamint a szerves kationtranszporter SLC22A4 és a RUNX1 gének intron SNP-i (Tokuhiro et al., 2003).
A fentiek mellett számos citokin például IL-1, IL-1 receptor antagonista, TNF- (Cvetkovic et al., 2002), TNF receptor (Maury et al., 2003), IFN- (Constantin et al., 2001), IL-3, IL-4 (Genevay et al., 2002), IL-6 (Pascual et al., 2000), IL-10, IL-18 (Sivalingam et al., 2003), IL-26 (Vandenbroeck et al., 2003), kemokinek (például RANTES, Makki et al., 2000), kemokin receptorok (CCR5, Zapico et al., 2000) és más proteinek génjeiben írtak le RA-szel összefüggő polimorfizmusokat.
Összefoglalás
A reumatoid artritisz korszerű kutatása, ami a patomechanizmus, a korai diagnosztika, a terápia céljait szolgálja ma és egyre inkább a közeljövőben a rendszerszemléletű biológián alapul.
A sikeres reumagenomika-kutatás ma a klinikus, a laboratóriumi szakember és a kutató (genetikus, immunológus, molekuláris biológus) együttműködő hálózatában valósul meg. A grid központjában a bioinformatikus áll, aki egyrészt az adatok (genetikai, laboratóriumi, klinikai) adatbázisokba foglalását végzi, másrészt adatelemzést folytat. Kiszolgálja ezt a rendszert a bibliográfia naprakész követése, amelyet ma már számítógépes elemzés is képes segíteni. A rendszer lényegi része a biobank, ahonnan a nagy kohortokon alapuló mintagyűjtemények származnak. Mindezek együtt képezik az integratív klinikai genomika körvonalait.
Jelenleg az integrativ genomikai analízis feltételei tehát:
1. Nagy (több ezer) mintaszám (biobankok nélkülözhetetlensége)
2. Nagyszámú (százezres nagyságrend) SNP meghatározása
3. Teljes genomot érintő eljárások (CGH, teljes genom expressziós és splice variáns analízisek) alkalmazása
4. Proteomikai és metabolomikai profilanalízis
5. Informatikai feldolgozás
Főbb ígéretek:
1. A reumatoid artritisz endofenotípusait is figyelembe vevő új SNP és expressziós mintázati profilok feltárása
2. Új biomarkerek kiemelése, új diagnosztikai eljárások céljából
3. Új gyógyszercélpontok kijelölése
Az összefoglaló szerzői biztosak abban, hogy a reumatológia modern prevenciós, diagnosztikai és terápiás irányzatai ma már elválaszthatatlanok a genomikától, és a bővülő helyi lehetőségek megfelelő kihasználása új perspektívát ad a hazai reumatológiai kutatásnak is.
Kulcsszavak: reumatológia, SNP, génexpresszió, genomika, patomechanizmus
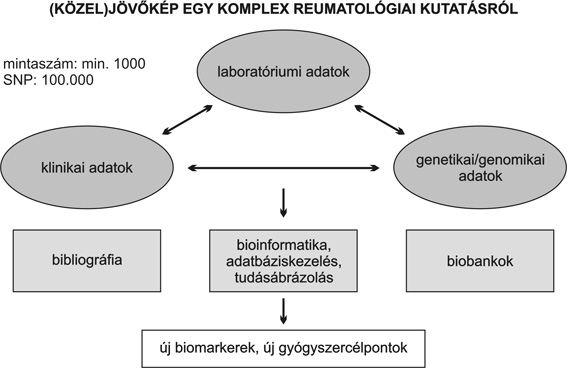
1. ábra • (Közel)jövőkép egy komplex reumatológiai kutatásról
Irodalom
Begovich, Ann B. – Carlton, V. E. – Honigberg, L. A. – Schrodi, S. J. – Chokkalingam, A. P. – Alexander, H. C. – Ardlie, K. G. – Huang, Q. – Smith, A. M. – Spoerke, J. M. – Conn, M. T. – Chang, M. – Chang, S. Y. – Saiki, R. K. – Catanese, J. J. – Leong, D. U. – Garcia, V. E. – McAllister, L. B. – Jeffery, D. A. – Lee, A. T. – Batliwalla, F. – Remmers, E. – Criswell, L. A. – Seldin, M. F. – Kastner, D. L. – Amos, C. I. – Sninsky, J. J. – Gregersen, P. K. (2004): A Missense Single-Nucleotide Polymorphism in a Gene Encoding a Protein Tyrosine Phosphatase (PTPN22) Is Associated with Rheumatoid Arthritis. The American Journal of Human Genetics.75, 330–337.
Chandonia, John-Marc – Brenner, Steven E. (2006): The Impact of Structural Genomics: Expectations and Outcomes. Review. Science. 311, 347–351.
Constantin, Arnaud – Navaux, F. – Lauwers- Cancčs, V. – Abbal, M. – van Meerwijk, J. P. – Mazičres, B. – Cambon-Thomsen, A. – Cantagrel, A. (2001): Interferon Gamma Gene Polymorphism and Susceptibility to, and Severity of, Rheumatoid Arthritis. The Lancet. 358, 2051–2052.
Cvetkovic, Jasmina T. – Wĺllberg-Jonsson, S. – Ahmed, E. – Rantapää-Dahlqvist, S. – Lefvert, A. K. (2002): Increased Levels of Autoantibodies Against Copper-Oxidized Low Density Lipoprotein, Malondialdehyde-Modified Low Density Lipoprotein and Cardiolipin in Patients with Rheumatoid Arthritis. Rheumatology (Oxford). 41, 988–995.
Genevay, Stephane – Di Giovine, F. S. – Perneger, T. V. – Silvestri, T. – Stingelin, S. – Duff, G. – Guerne, P. A. (2002): Association of Interleukin-4 and Interleukin-1B Gene Variants with Larsen Score Progression in Rheumatoid Arthritis. Arthritis and Rheumatism. 47, 303–309.
Gregersen, Peter K. – Silver, J. – Winchester, R. J. (1987): The shared epitope hypothesis. An Approach to Understanding the Molecular Genetics of Susceptibility to Rheumatoid Arthritis. Arthritis and Rheumatism. 30, 1205–1213.
Hall, Frances C. – Weeks, D. E. – Camilleri, J. P. – Williams, L. A. – Amos, N. – Darke, C. – Gibson, K. – Pile, K. – Wordsworth, B. P. – Jessop, J. D. (1996): Influence of the HLA-DRB1 Locus on Susceptibility and Severity in Rheumatoid Arthritis. QJM. 89, 821–829.
Ilonen, Jorma – Reijonen, H. – Arvilommi, H. – Jokinen, I. – Mottonen, T. – Hannonen, P. (1990): HLA-DR Antigens and HLA-DQ Beta Chain Polymorphism in Susceptibility to Rheumatoid Arthritis. Annals of the Rheumatic Diseases. 49, 494–496.
MacGregor, Alex J. – Snieder, H. – Schork, N. J. – Spector, T. D. (2000): Twins. Novel Uses to Study Complex Traits and Genetic Diseases. Trends in Genetics. 16,131–134.
Makki, R. F. – al Sharif, F. – González -Gay, M. A. – Garcia- Porrúa, C. – Ollier, W. E. – Hajeer, A. H. (2000): RANTES Gene Polymorphism in Polymyalgia Rheumatica, Giant Cell Arteritis and Rheumatoid Arthritis. Clinical and Experimental Rheumatology. 18, 391–393.
Maury, C. Peter – Liljestrom, M. – Laiho, K. – Tiitinen, S. – Kaarela, K. – Hurme, M. (2003): Tumor Necrosis Factor Alpha, Its Soluble Receptor I, and -308 Gene Promoter Polymorphism in Patients with Rheumatoid Arthritis with or without Amyloidosis: Implications for the Pathogenesis of Nephropathy and Anemia of Chronic Disease in Reactive Amyloidosis. Arthritis and Rheumatism. 48, 3068–3076.
Pascual, Maria – Nieto, A. – Matarán, L. – Balsa, A. – Pascual-Salcedo, D. – Martin, J. (2000): IL-6 Promoter Polymorphisms in Rheumatoid Arthritis. Genes and Immunity. 1, 338–340.
Sivalingam, S. P. – Yoon, K. H. – Koh, D. R. – Fong, K. Y. (2003): Single-Nucleotide Polymorphisms of the Interleukin-18 Gene Promoter Region in Rheumatoid Arthritis Patients: Protective Effect of AA Genotype. Tissue Antigens. 62, 498–504.
Suzuki, K. – Sawada, T. – Murakami, A. – Matsui, T. – Tohma, S. – Nakazono, K. – Takemura, M. – Takasaki, Y. – Mimori, T. – Yamamoto, K. (2003): High Diagnostic Performance of ELISA Detection of Antibodies to Citrullinated Antigens in Rheumatoid Arthritis. Scandinavian Journal of Rheumatology. 32, 197–204.
Tokuhiro, Shinya – Yamada, R. – Chang, X. – Suzuki, A. – Kochi, Y. – Sawada, T. – Suzuki, M. – Nagasaki, M. – Ohtsuki, M. – Ono, M. – Furukawa, H. – Nagashima, M. – Yoshino, S. – Mabuchi, A. – Sekine, A. – Saito, S. – Takahashi, A. – Tsunoda, T. – Nakamura, Y. – Yamamoto, K. (2003): An Intronic SNP in a RUNX1 Binding Site of SLC22A4, Encoding an Organic Cation Transporter, Is Associated with Rheumatoid Arthritis. Nature Genetics. 35, 341–348.
Vandenbroeck, Koen – Cunningham, S. – Goris, A. – Alloza, I. – Heggarty, S. – Graham, C. – Bell, A. – Rooney, M. (2003): Polymorphisms in the Interferon-Gamma/Interleukin-26 Gene Region Contribute to Sex Bias in Susceptibility to Rheumatoid Arthritis. Arthritis and Rheumatism. 48, 2773–2778.
Wordsworth, Paul – Halliday, Dorothy (2001): The Real Connective Tissue Diseases. Clinical Medicine. 1, 21–214.
Zapico, Isabel – Coto, E. – Rodriguez, A. – Ávarez, C. – Torre, J. C. – Álvarez, V. (2000): CCR5 (Chemokine Receptor-5) DNA-Polymorphism Influences the Severity of Rheumatoid Arthritis. Genes and Immunity. 1, 288–289.
<-- Vissza a 2006/10 szám tartalomjegyzékére
<-- Vissza a Magyar Tudomány honlapra
[Információk] [Tartalom] [Akaprint Kft.]